| nom | profession | |
|---|---|---|
| 0 | Astérix | |
| 1 | Obélix | Tailleur de menhir |
| 2 | Assurancetourix | Barde |
Nous avons vu dans les chapitres précédents comment récupérer, et harmoniser, des données issues de multiples sources: fichiers type CSV, API, webscraping, etc. Le panorama des manières possibles de consommer de la donnée serait incomplet sans évoquer un nouveau venu dans le paysage de la donnée, à savoir le format de données Parquet.
Du fait de ses caractéristiques techniques pensées pour l’analyse de données, et de sa simplicité d’usage avec Python, ce format devient de plus en plus incontournable. Il s’agit d’ailleurs d’une pierre angulaire des infrastructures cloud qui, depuis le milieu des années 2010, tendent à devenir l’environnement usuel dans le domaine de la data science (pour plus de détails, voir le cours de mise en production de Romain Avouac et moi).
- Comprendre les enjeux liés au stockage et au traitement de différents formats de données ;
- Distinguer le stockage sous forme de fichier et sous forme de base de données ;
- Découvrir le format
Parquet, ses avantages par rapport aux formats plats ou propriétaires ;
- Apprendre à traiter ces données avec
ArrowetDuckDB;
- Identifier les implications du stockage dans le cloud et comment
Pythonpeut s’y adapter.
Ce chapitre s’appuie sur un atelier dédié au sujet que j’ai donné dans le cadre du réseau des data scientists de la statistique publique (SSPHub)
Afficher les slides associées
Regarder le replay de la session live du 09 Avril 2025:
1 Elements de contexte
1.1 Principe du stockage de la donnée
Avant de comprendre les apports du format Parquet, il est utile de revenir brièvement sur la manière dont l’information est stockée et rendue accessible à un langage de traitement comme Python1.
Deux approches principales coexistent : le stockage sous forme de fichiers et celui sous forme de bases de données relationnelles. La distinction entre ces deux paradigmes repose sur la façon dont l’accès aux données est organisé.
1.2 Le stockage sous forme de fichiers
1.2.1 Les fichiers plats
Dans un fichier plat, les données sont organisées de manière linéaire, souvent séparées par un caractère (virgule, point-virgule, tabulation). Exemple avec un fichier .csv :
nom ; profession
Astérix ;
Obélix ; Tailleur de menhir ;
Assurancetourix ; BardePython peut facilement structurer cette information :
StringIOpermet de traiter la chaîne de caractère comme le contenu d’un fichier.
À propos des fichiers de ce type, on parle de fichiers plats car les enregistrements relatifs à une observation sont stockés ensemble, sans hiérarchie.
1.2.2 Les fichiers hiérarchiques
D’autres formats, comme JSON, structurent les données de manière hiérarchique :
[
{
"nom": "Astérix"
},
{
"nom": "Obélix",
"profession": "Tailleur de menhir"
},
{
"nom": "Assurancetourix",
"profession": "Barde"
}
]Cette fois, quand on n’a pas d’information, on ne se retrouve pas avec nos deux séparateurs accolés (cf. la ligne “Astérix”) mais l’information n’est tout simplement pas collectée.
La différence entre un fichier .csv et un fichier JSON ne réside pas seulement dans le format : elle implique une autre logique de stockage.
Le format JSON, non tabulaire, est plus souple : il permet de mettre à jour la structure des données sans recompiler ou modifier les anciennes lignes. Cela facilite la collecte évolutive dans des contextes comme les API.
Par exemple, un site web qui collecte de nouvelles données n’aura pas à mettre à jour l’ensemble de ses enregistrements antérieurs pour stocker la nouvelle donnée (par exemple pour indiquer que pour tel ou tel client cette donnée n’a pas été collectée) mais pourra la stocker dans un nouvel item.
Ce sera à l’outil de requête (Python ou un autre outil)
de créer une relation entre les enregistrements stockés à des endroits différents.
C’est ce principe qui sous-tend de nombreuses bases NoSQL (comme ElasticSearch), centrales dans l’univers du big data.
1.2.3 Données réparties sur plusieurs fichiers
Il est fréquent qu’une observation soit répartie entre plusieurs fichiers de formats différents. Par exemple, en géomatique, les contours géographiques peuvent être stockés de différentes manières pour accompagner les données qu’elles contextualisent:
- Soit tout est empilé dans un unique fichier qui contient à la fois les contours géographiques et les valeurs attributaires. Cette logique est celle suivie, par exemple, par le
GeoJSON; - Soit plusieurs fichiers se répartissent l’ensemble des données et, pour lire la donnée dans son ensemble (contours géographiques, données des différentes zones géographiques, système de projection, etc.), il faudra donc associer ceux-ci pour avoir un tableau de données complet. C’est l’approche suivie par le format
Shapefile.
Lorsque la donnée est éclatée dans plusieurs fichiers, c’est alors à l’outil de traitement (ex. Python) d’effectuer la jonction logique.
1.2.4 Le rôle du file system
Le système de fichiers (file system) permet à l’ordinateur de localiser physiquement les fichiers sur le disque. C’est un composant central dans la gestion de fichiers : il assure leur nommage, leur hiérarchie et leur accès.
1.3 Le stockage sous forme de bases de données
La logique des bases de données est différente. Elle est plus systémique. Une base de données relationnelle est gérée par un Système de Gestion de Base de Données (SGBD) qui permet :
- de stocker des ensembles cohérents de données,
- d’en permettre la mise à jour (ajout, suppression, modification),
- d’en contrôler l’accès (droits utilisateurs, types de requêtes, etc.).
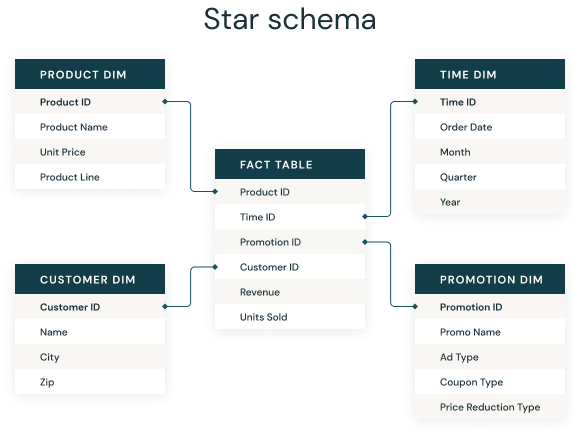
Les données sont organisées en tables reliées par des relations, souvent selon un schéma en étoile :
Le logiciel associé à la base de données fera ensuite le lien entre ces tables à partir de requêtes SQL. L’un des logiciels les plus efficaces dans ce domaine est PostgreSQL.
Python est tout à fait utilisable pour passer une requête SQL à un gestionnaire de base de données. Historiquement, les packages sqlalchemy et psycopg2 ont été très utilisés pour envoyer des requêtes à une base de données PostgreSQL (lire celle-ci, la mettre à jour, etc.). Aujourd’hui, DuckDB, sur lequel nous reviendrons lorsque nous parlerons du format Parquet, est un choix pratique pour passer des requêtes SQL à un SGBD PostgreSQL.
Pourquoi les fichiers ont le vent en poupe
Le succès croissant des fichiers dans l’écosystème de la data science s’explique par plusieurs facteurs techniques et pratiques qui les rendent particulièrement adaptés aux usages analytiques modernes.
En premier lieu, les fichiers sont beaucoup plus légers à manipuler que les bases de données. Ils ne nécessitent pas l’installation ou le maintien d’un logiciel de gestion spécialisé : un simple file system, déjà présent sur tout système d’exploitation, suffit à y accéder.
Pour lire un fichier dans Python, il suffit d’utiliser une librairie comme Pandas. À l’inverse, interagir avec une base de données implique souvent :
- l’installation et la configuration d’un SGBD (comme
PostgreSQL,MySQL, etc.) ; - la gestion d’une connexion réseau ;
- le recours à des bibliothèques comme
sqlalchemyoupsycopg2.
Cette différence de complexité rend l’approche fichier beaucoup plus souple et rapide pour les tâches exploratoires ou ponctuelles.
Cette légèreté a une contrepartie : les fichiers ne permettent pas une gestion fine des droits d’accès. Il est difficile, par exemple, d’empêcher un utilisateur de modifier ou supprimer la donnée à moins de dupliquer le fichier et de travailler sur une copie. C’est l’une des limites de l’approche fichier dans les environnements multi-utilisateurs mais auxquelles les solutions cloud, notamment la technologie S3 sur laquelle nous reviendrons, apportent des réponses.
La principale raison pour laquelle les fichiers sont souvent privilégiés par rapport aux SGBD réside dans la nature des opérations effectuées. Les bases de données relationnelles prennent tout leur sens lorsque l’on doit gérer des écritures fréquentes ou des mises à jour complexes sur des ensembles de données structurés — c’est-à-dire dans une logique applicative, où la donnée évolue continuellement (ajout, modification, suppression).
À l’inverse, dans un contexte analytique, on se contente généralement de lire et de manipuler temporairement des données sans modifier la source. L’objectif est d’interroger, d’agréger, de filtrer — pas de pérenniser les changements. Pour ce type d’usage, les fichiers (notamment dans des formats optimisés comme Parquet, comme nous allons le voir) sont parfaitement adaptés : ils offrent une lecture rapide, une portabilité élevée et n’imposent pas l’intermédiation d’un moteur de base de données.
2 Le format Parquet
Le format CSV a longtemps été plébiscité en raison de sa simplicité :
- Il est lisible par un humain (un simple éditeur de texte suffit pour en lire le contenu) ;
- Il repose sur une structure tabulaire simple, bien adaptée à de nombreuses situations d’analyse ;
- Il est universel et interopérable, car non dépendant d’un logiciel particulier.
Mais cette simplicité a un coût. Plusieurs limites du format CSV ont justifié l’émergence de formats plus performants pour l’analyse de données comme Parquet
2.1 Limites du format CSV
Le CSV est un format lourd :
- Il n’est pas compressé, ce qui augmente sa taille disque ;
- Toutes les données y sont stockées de façon brute. L’optimisation du typage (entier, flottant, chaîne…) est laissée à la librairie qui l’importe (comme
Pandas), ce qui nécessite de scanner les données à l’ouverture, augmentant le temps de chargement et le risque d’erreur.
Le CSV est orienté ligne :
- Pour accéder à une colonne spécifique, il faut lire chaque ligne du fichier puis en extraire la colonne d’intérêt ;
- Ce modèle est peu performant lorsqu’on souhaite ne manipuler qu’un sous-ensemble de colonnes — un cas très courant en data science.
Le CSV est coûteux à modifier :
Ajouter une colonne ou insérer une donnée intermédiaire implique de réécrire tout le fichier. Par exemple, ajouter une colonne
cheveuxnécessiterait de produire une nouvelle version du fichier :nom ; cheveux ; profession Astérix ; blond ; Obélix ; roux ; Tailleur de menhir Assurancetourix ; blond ; Barde
La plupart des outils de data science proposent des formats de sérialisation spécifiques :
.picklepourPython,
.rdaou.RDatapourR,
.dtapourStata,
.sas7bdatpourSAS.
Cependant, ces formats sont propriétaires ou fortement couplés à un langage, ce qui pose des problèmes d’interopérabilité. Par exemple, Python ne peut pas lire nativement un .sas7bdat. Même s’il existe des bibliothèques dédiées, l’absence de documentation officielle rend le support incertain.
À ce titre, malgré ses limites, le .csv conserve l’avantage de l’universalité. Mais le format Parquet combine cette portabilité avec des performances bien supérieures.
2.2 L’émergence du format Parquet
Pour répondre à ces limites, le format Parquet, développé comme projet open-source Apache, propose une approche radicalement différente.
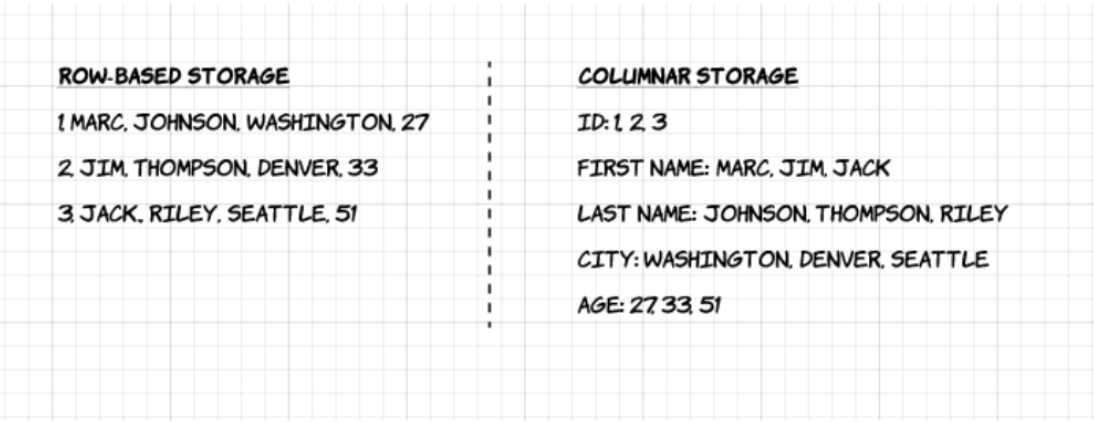
Sa principale caractéristique : il est orienté colonne. Contrairement au CSV, les données de chaque colonne sont stockées séparément. Cela permet :
- de charger uniquement les colonnes utiles à une analyse ;
- de compresser plus efficacement les données ;
- d’accélérer significativement les requêtes sélectives.
Voici une représentation tirée du blog d’Upsolver qui illustre la différence entre stockage ligne (row-based) et stockage colonne (columnar) :
Dans notre exemple, on pourrait lire la colonne profession sans parcourir les noms, ce qui rend l’accès plus rapide (ignorez l’élément pyarrow.Table, nous
reviendrons dessus) :
pyarrow.Table
nom : string
profession: string
----
nom : [["Astérix ","Obélix ","Assurancetourix "]]
profession: [["","Tailleur de menhir","Barde"]]Grâce à la structure orientée colonne, il est possible de lire uniquement une variable (comme profession) sans avoir à parcourir toutes les lignes du fichier.
path
└── to
└── table
├── gender=male
│ ├── country=US
│ │ └── data.parquet
│ ├── country=CN
│ │ └── data.parquet
└── gender=female
├── country=US
│ └── data.parquet
├── country=CN
│ └── data.parquetÀ la lecture, l’ensemble est reconstruit sous forme tabulaire :
root
|-- name: string (nullable = true)
|-- age: long (nullable = true)
|-- gender: string (nullable = true)
|-- country: string (nullable = true)2.3 Un format taillé pour l’analyse - pas uniquement le big data
Comme le rappelle le blog d’Upsolver :
Complex data such as logs and event streams would need to be represented as a table with hundreds or thousands of columns, and many millions of rows. Storing this table in a row-based format such as CSV would mean:
- Queries will take longer to run since more data needs to be scanned…
- Storage will be more costly since CSVs are not compressed as efficiently as Parquet
Mais le format Parquet n’est pas réservé aux architectures big data. Toute personne produisant ou manipulant des jeux de données bénéficiera de ses qualités :
- fichiers plus petits,
- import rapide et fiable,
2.4 Lire un Parquet en Python: exemple
Il existe de nombreuses librairies fonctionnant bien avec Parquet mais les deux plus utiles à connaître sont PyArrow et DuckDB. Nous avons déjà évoqué succinctement celles-ci lorsqu’il était question des alternatives à Pandas gérant mieux les données volumineuses. Ces librairies peuvent servir à effectuer les premières opérations lourdes avant de convertir les données obtenues, plus légères, en pd.DataFrame.
La librairie PyArrow permet de lire et écrire des fichiers Parquet tout en tirant parti de la structure colonne du format2. Elle repose sur un objet pyarrow.Table, qui peut, une fois les calculs lourds effectués, être converti vers un DataFrame Pandas pour bénéficier d’un écosystème plus riche en fonctionnalités.
La librairie DuckDB permet d’interroger directement des fichiers Parquet à l’aide du langage SQL, sans les charger entièrement en mémoire. Autrement dit, elle reprend la philosophie du monde de la base de données (l’utilisation de SQL) mais sur des fichiers. Le résultat des requêtes peut, là aussi, être converti en DataFrame Pandas, ce qui permet de profiter à la fois de la souplesse de Pandas et de la performance du moteur SQL embarqué. Fonctionnalité moins connue, cette librairie permet aussi d’effectuer des opérations SQL directement sur un DataFrame Pandas. Ceci peut être pertinent pour des situations où la syntaxe Pandas est peu pratique là où SQL est très bien fait ; par exemple, pour créer une nouvelle variable comme le résultat d’une statistique par groupe.
L’utilisation des alias pa pour pyarrow et pq pour pyarrow.parquet est une convention largement adoptée, à l’image de celle de pd pour pandas.
Pour illustrer ces fonctionnalités, prenons un jeu de données issu des données synthétiques du recensement de la population diffusés par l’Insee.
import requests
import pyarrow.parquet as pq
# Example Parquet
url = "https://minio.lab.sspcloud.fr/projet-formation/bonnes-pratiques/data/RPindividus/REGION=93/part-0.parquet"
# Télécharger le fichier et l'enregistrer en local
with open("example.parquet", "wb") as f:
response = requests.get(url)
f.write(response.content)L’idéal pour bénéficier pleinement des optimisations permises par le format Parquet est de passer par pyarrow.dataset. Cela permettra de bénéficier des optimisations permises par le combo Parquet et Arrow, que toutes les manières de lire un Parquet avec Arrow ne proposent pas (cf. prochains exercices).
import pyarrow.dataset as ds
dataset = ds.dataset(
"example.parquet"
).scanner(columns = ["AGED", "IPONDI", "DEPT"])
table = dataset.to_table()
tablepyarrow.Table
AGED: int32
IPONDI: double
DEPT: dictionary<values=string, indices=int32, ordered=0>
----
AGED: [[9,12,40,70,52,...,29,66,72,75,77],[46,76,46,32,2,...,7,5,37,29,4],...,[67,37,45,56,75,...,64,37,47,20,18],[16,25,51,6,11,...,93,90,92,21,65]]
IPONDI: [[2.73018871840726,2.73018871840726,2.73018871840726,0.954760150327854,3.75907197064638,...,3.27143319621654,4.83980378599556,4.83980378599556,4.83980378599556,4.83980378599556],[3.02627578376137,3.01215358930406,3.01215358930406,2.93136309038958,2.93136309038958,...,2.96848755763453,2.96848755763453,3.25812879950072,3.25812879950072,1.12514509319438],...,[2.57931132917563,2.85579410739065,0.845993555838931,2.50296716736141,3.70786113613679,...,3.08375347880892,2.88038807573222,3.22776230929947,3.22776230929947,3.22776230929947],[3.22776230929947,3.22776230929947,3.22776230929947,3.29380242174036,3.29380242174036,...,5.00000768518755,5.00000768518755,5.00000768518755,5.00000768518755,1.00000153703751]]
DEPT: [ -- dictionary:
["01","02","03","04","05",...,"95","971","972","973","974"] -- indices:
[5,5,5,5,5,...,5,5,5,5,5], -- dictionary:
["01","02","03","04","05",...,"95","971","972","973","974"] -- indices:
[5,5,5,5,5,...,5,5,5,5,5],..., -- dictionary:
["01","02","03","04","05",...,"95","971","972","973","974"] -- indices:
[84,84,84,84,84,...,84,84,84,84,84], -- dictionary:
["01","02","03","04","05",...,"95","971","972","973","974"] -- indices:
[84,84,84,84,84,...,84,84,84,84,84]]Pour importer et traiter ces données, on peut conserver
les données sous le format pyarrow.Table
ou transformer en pandas.DataFrame. La deuxième
option est plus lente mais présente l’avantage
de permettre ensuite d’appliquer toutes les
manipulations offertes par l’écosystème
pandas qui est généralement mieux connu que
celui d’Arrow.
import duckdb
duckdb.sql("""
FROM read_parquet('example.parquet')
SELECT AGED, IPONDI, DEPT
""")┌───────┬───────────────────┬─────────┐
│ AGED │ IPONDI │ DEPT │
│ int32 │ double │ varchar │
├───────┼───────────────────┼─────────┤
│ 9 │ 2.73018871840726 │ 06 │
│ 12 │ 2.73018871840726 │ 06 │
│ 40 │ 2.73018871840726 │ 06 │
│ 70 │ 0.954760150327854 │ 06 │
│ 52 │ 3.75907197064638 │ 06 │
│ 82 │ 3.21622922493506 │ 06 │
│ 6 │ 3.44170061276923 │ 06 │
│ 12 │ 3.44170061276923 │ 06 │
│ 15 │ 3.44170061276923 │ 06 │
│ 43 │ 3.44170061276923 │ 06 │
│ · │ · │ · │
│ · │ · │ · │
│ · │ · │ · │
│ 68 │ 2.73018871840726 │ 06 │
│ 35 │ 3.46310256220757 │ 06 │
│ 2 │ 3.46310256220757 │ 06 │
│ 37 │ 3.46310256220757 │ 06 │
│ 84 │ 3.69787960424482 │ 06 │
│ 81 │ 4.7717265388427 │ 06 │
│ 81 │ 4.7717265388427 │ 06 │
│ 51 │ 3.60566450823737 │ 06 │
│ 25 │ 3.60566450823737 │ 06 │
│ 13 │ 3.60566450823737 │ 06 │
├───────┴───────────────────┴─────────┤
│ ? rows 3 columns │
│ (>9999 rows, 20 shown) │
└─────────────────────────────────────┘2.5 Des exercices pour en apprendre plus
Voici une série d’exercices issues du cours de mise en production de projets data science que Romain Avouac et moi proposons à la fin du cursus d’ingénieurs de l’ENSAE.
Ces exercices illustrent progressivement quelques concepts présentés ci-dessus tout en présentant les bonnes pratiques à adopter pour traiter des données volumineuses. La correction de ces exercices est disponible sur la page du cours en question.
Tout au long de cette application, nous allons voir comment utiliser le format Parquet de la manière la plus efficiente possible. Afin de comparer les différents formats et méthodes d’utilisation, nous allons comparer le temps d’exécution et l’usage mémoire d’une requête standard. Commençons déjà, sur un premier exemple avec une donnée légère, pour comparer les formats CSV et Parquet.
Pour cela, nous allons avoir besoin de récupérer des données au format Parquet. Nous proposons d’utiliser les données détaillées et anonymisées du recensement de la population française: environ 20 millions de lignes pour 80 colonnes. Le code pour récupérer celles-ci est donné ci-dessous
Code pour récupérer les données
import pyarrow.parquet as pq
import pyarrow as pa
import os
# Définir le fichier de destination
filename_table_individu = "data/RPindividus.parquet"
# Copier le fichier depuis le stockage distant (remplacer par une méthode adaptée si nécessaire)
1os.system("mc cp s3/projet-formation/bonnes-pratiques/data/RPindividus.parquet data/RPindividus.parquet")
# Charger le fichier Parquet
table = pq.read_table(filename_table_individu)
df = table.to_pandas()
# Filtrer les données pour REGION == "24"
df_filtered = df.loc[df["REGION"] == "24"]
# Sauvegarder en CSV
df_filtered.to_csv("data/RPindividus_24.csv", index=False)
# Sauvegarder en Parquet
pq.write_table(pa.Table.from_pandas(df_filtered), "data/RPindividus_24.parquet")- 1
-
Cette ligne de code utilise l’utilitaire Minio Client disponible sur le
SSPCloud. Si vous n’êtes pas sur cette infrastructure, vous pouvez vous référer à la boite dédiée
Vous devrez remplacer la ligne
os.system("mc cp s3/projet-formation/bonnes-pratiques/data/RPindividus.parquet data/RPindividus.parquet")qui utilise l’outil en ligne de commande mc par un code téléchargeant cette donnée à partir de l’URL https://projet-formation.minio.lab.sspcloud.fr/bonnes-pratiques/data/RPindividus.parquet.
Il y a de nombreuses manières de faire. Vous pouvez par exemple le faire en pur Python avec requests. Si vous avez curl installé, vous pouvez aussi l’utiliser. Par l’intermédiaire de Python, cela donnera la commande os.system("curl -o data/RPindividus.parquet https://projet-formation/bonnes-pratiques/data/RPindividus.parquet").
Ces exercices vont utiliser des décorateurs Python, c’est-à-dire des fonctions qui surchargent le comportement d’une autre fonction. En l’occurrence, nous allons créer une fonction exécutant une chaîne d’opérations et la surcharger avec une autre chargée de contrôler l’usage mémoire et le temps d’exécution.
- Créer un notebook
benchmark_parquet.ipynbafin de réaliser les différentes comparaisons de performance de l’application - Créons notre décorateur, en charge de benchmarker le code
Python:
Dérouler pour retrouver le code du décorateur permettant de mesurer la performance
::: {#006cd4ef .cell execution_count=8} ``` {.python .cell-code} import time from memory_profiler import memory_usage from functools import wraps import warnings
def convert_size(size_bytes):
if size_bytes == 0:
return "0B"
size_name = ("B", "KB", "MB", "GB", "TB", "PB", "EB", "ZB", "YB")
i = int(math.floor(math.log(size_bytes, 1024)))
p = math.pow(1024, i)
s = round(size_bytes / p, 2)
return "%s %s" % (s, size_name[i])
# Decorator to measure execution time and memory usage
def measure_performance(func, return_output=False):
@wraps(func)
def wrapper(return_output=False, *args, **kwargs):
warnings.filterwarnings("ignore")
start_time = time.time()
mem_usage = memory_usage((func, args, kwargs), interval=0.1)
end_time = time.time()
warnings.filterwarnings("always")
exec_time = end_time - start_time
peak_mem = max(mem_usage) # Peak memory usage
exec_time_formatted = f"\033[92m{exec_time:.4f} sec\033[0m"
peak_mem_formatted = f"\033[92m{convert_size(1024*peak_mem)}\033[0m"
print(f"{func.__name__} - Execution Time: {exec_time_formatted} | Peak Memory Usage: {peak_mem_formatted}")
if return_output is True:
return func(*args, **kwargs)
return wrapper:::
</details>
* Reprendre ce code pour encapsuler un code de construction d'une pyramide des âges dans une fonction `process_csv_appli1`
<details>
<summary>
Dérouler pour récupérer le code pour mesurer les performances de la lecture en CSV
</summary>
::: {#841d0529 .cell execution_count=9}
``` {.python .cell-code}
# Apply the decorator to functions
@measure_performance
def process_csv_appli1(*args, **kwargs):
df = pd.read_csv("data/RPindividus_24.csv")
return (
df.loc[df["DEPT"] == 36]
.groupby(["AGED", "DEPT"])["IPONDI"]
.sum().reset_index()
.rename(columns={"IPONDI": "n_indiv"})
):::
Exécuter
process_csv_appli1()etprocess_csv_appli1(return_output=True)Sur le même modèle, construire une fonction
process_parquet_appli1basée cette fois sur le fichierdata/RPindividus_24.parquetchargé avec la fonction read_parquet dePandasComparer les performances (temps d’exécution et allocation mémoire) de ces deux méthodes grâce à la fonction.
Correction complète
import math
import pandas as pd
import time
from memory_profiler import memory_usage
from functools import wraps
import warnings
def convert_size(size_bytes):
if size_bytes == 0:
return "0B"
size_name = ("B", "KB", "MB", "GB", "TB", "PB", "EB", "ZB", "YB")
i = int(math.floor(math.log(size_bytes, 1024)))
p = math.pow(1024, i)
s = round(size_bytes / p, 2)
return "%s %s" % (s, size_name[i])
# Decorator to measure execution time and memory usage
def measure_performance(func, return_output=False):
@wraps(func)
def wrapper(return_output=False, *args, **kwargs):
warnings.filterwarnings("ignore")
start_time = time.time()
mem_usage = memory_usage((func, args, kwargs), interval=0.1)
end_time = time.time()
warnings.filterwarnings("always")
exec_time = end_time - start_time
peak_mem = max(mem_usage) # Peak memory usage
exec_time_formatted = f"\033[92m{exec_time:.4f} sec\033[0m"
peak_mem_formatted = f"\033[92m{convert_size(1024*peak_mem)}\033[0m"
print(f"{func.__name__} - Execution Time: {exec_time_formatted} | Peak Memory Usage: {peak_mem_formatted}")
if return_output is True:
return func(*args, **kwargs)
return wrapper
# Apply the decorator to functions
@measure_performance
def process_csv(*args, **kwargs):
df = pd.read_csv("data/RPindividus_24.csv")
return (
df.loc[df["DEPT"] == 36]
.groupby(["AGED", "DEPT"])["IPONDI"]
.sum().reset_index()
.rename(columns={"IPONDI": "n_indiv"})
)
@measure_performance
def process_parquet(*args, **kwargs):
df = pd.read_parquet("data/RPindividus_24.parquet")
return (
df.loc[df["DEPT"] == "36"]
.groupby(["AGED", "DEPT"])["IPONDI"]
.sum().reset_index()
.rename(columns={"IPONDI": "n_indiv"})
)
process_csv()
process_parquet()❓️ Quelle semble être la limite de la fonction read_parquet ?
On gagne déjà un temps conséquent en lecture mais on ne bénéficie pas vraiment de l’optimisation permise par Parquet car on transforme les données directement après la lecture en DataFrame Pandas. On n’utilise donc pas l’une des fonctionnalités principales du format Parquet, qui explique ses excellentes performances: le predicate pushdown qui consiste à optimiser notre traitement pour faire remonter, le plus tôt possible, les filtres sur les colonnes pour ne garder que celles vraiment utilisées dans le traitement.
La partie précédente a montré un gain de temps considérable du passage de CSV à Parquet. Néanmoins, l’utilisation mémoire était encore très élevée alors qu’on utilise de fait qu’une infime partie du fichier.
Dans cette partie, on va voir comment utiliser la lazy evaluation et les optimisations du plan d’exécution effectuées par Arrow pour exploiter pleinement la puissance du format Parquet.
- Ouvrir le fichier
data/RPindividus_24.parquetavec pyarrow.dataset. Regarder la classe de l’objet obtenu. - Tester le code ci-dessous pour lire un échantillon de données:
(
dataset.scanner()
.head(5)
.to_pandas()
)Comprenez-vous la différence avec précédemment ? Observez dans la documentation la méthode to_table : comprenez-vous son principe ?
- Construire une fonction
summarize_parquet_arrow(resp.summarize_parquet_duckdb) qui importe cette fois les données avec la fonctionpyarrow.dataset(resp. avecDuckDB) et effectue l’agrégation voulue. - Comparer les performances (temps d’exécution et allocation mémoire) des trois méthodes (
Parquetlu et processé avecPandas,ArrowetDuckDB) grâce à notre fonction.
Correction
Code complet de l’application
import duckdb
import pyarrow.dataset as ds
@measure_performance
def summarize_parquet_duckdb(*args, **kwargs):
con = duckdb.connect(":memory:")
query = """
FROM read_parquet('data/RPindividus_24.parquet')
SELECT AGED, DEPT, SUM(IPONDI) AS n_indiv
GROUP BY AGED, DEPT
"""
return (con.sql(query).to_df())
@measure_performance
def summarize_parquet_arrow(*args, **kwargs):
dataset = ds.dataset("data/RPindividus_24.parquet", format="parquet")
table = dataset.to_table()
grouped_table = (
table
.group_by(["AGED", "DEPT"])
.aggregate([("IPONDI", "sum")])
.rename_columns(["AGED", "DEPT", "n_indiv"])
.to_pandas()
)
return (
grouped_table
)
process_parquet()
summarize_parquet_duckdb()
summarize_parquet_arrow()Avec l’évaluation différée, on obtient donc un processus en plusieurs temps:
ArrowouDuckDBreçoit des instructions, les optimise, exécute les requêtes- Seules les données en sortie de cette chaîne sont renvoyées à
Python
Ajoutez une étape de filtre sur les lignes dans nos requêtes:
- Avec
DuckDB, vous devez modifier la requête avec unWHERE DEPT IN ('18', '28', '36') - Avec
Arrow, vous devez modifier l’étapeto_tablede cette manière:dataset.to_table(filter=pc.field("DEPT").isin(['18', '28', '36']))
Correction
import pyarrow.dataset as ds
import pyarrow.compute as pc
import duckdb
@measure_performance
def summarize_filter_parquet_arrow(*args, **kwargs):
dataset = ds.dataset("data/RPindividus.parquet", format="parquet")
table = dataset.to_table(filter=pc.field("DEPT").isin(['18', '28', '36']))
grouped_table = (
table
.group_by(["AGED", "DEPT"])
.aggregate([("IPONDI", "sum")])
.rename_columns(["AGED", "DEPT", "n_indiv"])
.to_pandas()
)
return (
grouped_table
)
@measure_performance
def summarize_filter_parquet_duckdb(*args, **kwargs):
con = duckdb.connect(":memory:")
query = """
FROM read_parquet('data/RPindividus_24.parquet')
SELECT AGED, DEPT, SUM(IPONDI) AS n_indiv
WHERE DEPT IN ('11','31','34')
GROUP BY AGED, DEPT
"""
return (con.sql(query).to_df())
summarize_filter_parquet_arrow()
summarize_filter_parquet_duckdb()❓️ Pourquoi ne gagne-t-on pas de temps avec nos filtres sur les lignes (voire pourquoi en perdons nous?) comme c’est le cas avec les filtres sur les colonnes ?
La donnée n’est pas organisée par blocs de lignes comme elle l’est par bloc de colonne. Heureusement, il existe pour cela un moyen: le partitionnement !
La lazy evaluation et les optimisations d’Arrow apportent des gain de performance considérables. Mais on peut encore faire mieux ! Lorsqu’on sait qu’on va être amené à filter régulièrement les données selon une variable d’intérêt, on a tout intérêt à partitionner le fichier Parquet selon cette variable.
Parcourir la documentation de la fonction
pyarrow.parquet.write_to_datasetpour comprendre comment spécifier une clé de partitionnement lors de l’écriture d’un fichierParquet. Plusieurs méthodes sont possibles.Importer la table complète des individus du recensement depuis
"data/RPindividus.parquet"avec la fonctionpyarrow.dataset.datasetet l’exporter en une table partitionnée"data/RPindividus_partitionne.parquet", partitionnée par la région (REGION) et le département (DEPT).Observer l’arborescence des fichiers de la table exportée pour voir comment la partition a été appliquée.
Modifier nos fonctions d’import, filtre et agrégations via
ArrowouDuckDBpour utiliser, cette fois, leParquetpartitionné. Comparer à l’utilisation du fichier non partitionné.
Correction de la question 2 (écriture du Parquet partitionné)
import pyarrow.parquet as pq
dataset = ds.dataset(
"data/RPindividus.parquet", format="parquet"
).to_table()
pq.write_to_dataset(
dataset,
root_path="data/RPindividus_partitionne",
partition_cols=["REGION", "DEPT"]
)Correction de la question 4 (lecture du Parquet partitionné)
import pyarrow.dataset as ds
import pyarrow.compute as pc
import duckdb
@measure_performance
def summarize_filter_parquet_partitioned_arrow(*args, **kwargs):
dataset = ds.dataset("data/RPindividus_partitionne/", partitioning="hive")
table = dataset.to_table(filter=pc.field("DEPT").isin(['18', '28', '36']))
grouped_table = (
table
.group_by(["AGED", "DEPT"])
.aggregate([("IPONDI", "sum")])
.rename_columns(["AGED", "DEPT", "n_indiv"])
.to_pandas()
)
return (
grouped_table
)
@measure_performance
def summarize_filter_parquet_complete_arrow(*args, **kwargs):
dataset = ds.dataset("data/RPindividus.parquet")
table = dataset.to_table(filter=pc.field("DEPT").isin(['18', '28', '36']))
grouped_table = (
table
.group_by(["AGED", "DEPT"])
.aggregate([("IPONDI", "sum")])
.rename_columns(["AGED", "DEPT", "n_indiv"])
.to_pandas()
)
return (
grouped_table
)
@measure_performance
def summarize_filter_parquet_complete_duckdb(*args, **kwargs):
con = duckdb.connect(":memory:")
query = """
FROM read_parquet('data/RPindividus.parquet')
SELECT AGED, DEPT, SUM(IPONDI) AS n_indiv
WHERE DEPT IN ('11','31','34')
GROUP BY AGED, DEPT
"""
return (con.sql(query).to_df())
@measure_performance
def summarize_filter_parquet_partitioned_duckdb(*args, **kwargs):
con = duckdb.connect(":memory:")
query = """
FROM read_parquet('data/RPindividus_partitionne/**/*.parquet', hive_partitioning = True)
SELECT AGED, DEPT, SUM(IPONDI) AS n_indiv
WHERE DEPT IN ('11','31','34')
GROUP BY AGED, DEPT
"""
return (con.sql(query).to_df())
summarize_filter_parquet_complete_arrow()
summarize_filter_parquet_partitioned_arrow()
summarize_filter_parquet_complete_duckdb()
summarize_filter_parquet_partitioned_duckdb()❓️ Dans le cadre d’une mise à disposition de données en Parquet, comment bien choisir la/les clé(s) de partitionnement ? Quelle est la limite à garder en tête ?
2.6 Pour aller plus loin
- La formation aux bonnes pratiques
RetGitdéveloppée par l’Insee avec des éléments très similaires à ceux présentés dans ce chapitre. - Un atelier sur le format
Parquetet l’écosystèmeDuckDBpour l’EHESS avec des exemplesRetPythonutilisant la même source de données que l’application. - Le guide de prise en main des données du recensement au format
Parquetavec des exemples d’utilisation deDuckDBen WASM (directement depuis le navigateur, sans installationRouPython)
3 Les données dans le cloud
Le stockage cloud, dans le contexte de la science des données, reprend le principe de services comme Dropbox ou Google Drive : un utilisateur accède à des fichiers distants comme s’ils étaient sur son propre disque3. Autrement dit, pour un utilisateur de Python, la manipulation de fichiers stockés dans le cloud peut sembler identique à celle de fichiers locaux.
Mais contrairement à un dossier de type Mes Documents/monsuperfichier, les fichiers ne résident pas sur l’ordinateur local. Ils sont hébergés sur un serveur distant, et chaque opération (lecture, écriture) passe par une connexion réseau.
3.1 Pourquoi ne pas utiliser Dropbox ou Drive ?
Néanmoins, Dropbox ou Drive ne sont pas faits pour du stockage de données. Pour ces dernières, il est plus pertinent d’utiliser une technologie adaptée (voir le cours de mise en production). Les principaux fournisseurs de service cloud (AWS, GCP, Azure…) reposent sur le même principe avec un stockage orienté objet reposant sur une technologie de type S3.
C’est pourquoi les principaux fournisseurs cloud (AWS, Google Cloud, Azure…) proposent des solutions spécifiques au stockage de données, souvent basées sur des systèmes orientés objet, dont le plus connu est S3.
3.2 Le système S3
Le système S3 (Simple Storage Service), développé par Amazon, est devenu un standard dans le monde du stockage cloud. Il s’agit d’un système :
- fiable (réplication des données) ;
- sécurisé (données chiffrées, contrôle d’accès granulaire) ;
- scalable (adapté à des volumes massifs).
L’unité centrale de S3 est le bucket : un espace de stockage (privé ou public) qui peut contenir une arborescence de fichiers.
Pour accéder à un fichier dans un bucket :
- L’utilisateur doit être autorisé (via des identifiants ou des jetons d’accès, souvent appelés tokens) ;
- Une fois authentifié, il peut lire, écrire ou modifier les fichiers à l’intérieur du bucket, à la manière d’un système de fichiers distant.
3.4 Cas pratique : stocker les données de son projet sur le SSP Cloud
Une composante essentielle de l’évaluation des projets Python est la reproductibilité, i.e. la possibilité de retrouver les mêmes résultats à partir des mêmes données d’entrée et du même code. Dans la mesure du possible, il faut donc que votre rendu final parte des données brutes utilisées comme source dans votre projet. Si les fichiers de données source sont accessibles via une URL publique par exemple, il est idéal de les importer directement à partir de cette URL au début de votre projet (voir le TP Pandas pour un exemple d’un tel import via Pandas).
En pratique, cela n’est pas toujours possible. Peut-être que vos données ne sont pas directement publiquement accessibles, ou bien sont disponibles sous des formats complexes qui demandent des pré-traitements avant d’être exploitables dans un format de donnée standard. Peut-être que vos données résultent d’une phase de récupération automatisée via une API ou du webscraping, auquel cas l’étape de récupération peut prendre du temps à reproduire. Par ailleurs, les sites internet évoluent fréquemment dans le temps, il est donc préférable de “figer” les données une fois l’étape de récupération effectuée. De la même façon, même s’il ne s’agit pas de données source, vous pouvez vouloir entraîner des modèles et stocker leur version entraînée, car cette étape peut également être chronophage.
Dans toutes ces situations, il est nécessaire de pouvoir stocker des données (ou des modèles). Votre dépôt Git n’est pas le lieu adapté pour le stockage de fichiers volumineux. Un projet Python bien construit est modulaire: il sépare le stockage du code (Git), d’éléments de configuration (par exemple des jetons d’API qui ne doivent pas être dans le code) et du stockage des données. Cette séparation conceptuelle entre code et données permet de meilleurs projets.
Là où Git est fait pour stocker du code, on utilise des solutions adaptées pour le stockage de fichiers. De nombreuses solutions existent pour ce faire. Sur le SSP Cloud, on propose MinIO, une implémentation open-source du stockage S3 présenté plus haut. Ce court tutoriel vise à présenter une utilisation standard dans le cadre de vos projets.
Quelle que soit la solution de stockage retenue pour vos données/modèles, le code ayant servir à produire ces objets doit impérativement figurer dans votre dépôt de projet.
3.4.1 Partager des fichiers sur le SSP Cloud
Comme expliqué plus haut, on stocke les fichiers sur S3 dans un bucket. Sur le SSP Cloud, un bucket est créé automatiquement lors de votre création de compte, avec le même nom que votre compte SSP Cloud. L’interface Mes Fichiers vous permet d’y accéder de manière visuelle, d’y importer des fichiers, de les télécharger, etc.
Dans ce tutoriel, nous allons plutôt y accéder de manière programmatique, via du code Python. Le package s3fs permet de requêter votre bucket à la manière d’un filesystem classique. Par exemple, vous pouvez lister les fichiers disponibles sur votre bucket avec la commande suivante :
import s3fs
fs = s3fs.S3FileSystem(client_kwargs={"endpoint_url": "https://minio.lab.sspcloud.fr"})
MY_BUCKET = "mon_nom_utilisateur_sspcloud"
fs.ls(MY_BUCKET)Si vous n’avez jamais ajouté de fichier sur MinIO, votre bucket est vide, cette commande devrait donc renvoyer une liste vide. On va donc ajouter un premier dossier pour voir la différence.
Par défaut, un bucket vous est personnel, c’est à dire que les données qui s’y trouvent ne peuvent être lues ou modifiées que par vous. Dans le cadre de votre projet, vous aurez envie de partager ces fichiers avec les membres de votre groupe pour développer de manière collaborative. Mais pas seulement ! Il faudra également que vos correcteurs puissent accéder à ces fichiers pour reproduire vos analyses.
Il existe différentes possibilités de rendre des fichiers plus ou moins publics sur MinIO. La plus simple, et celle que nous vous recommandons, est de créer un dossier diffusion à la racine de votre bucket. Sur le SSP Cloud, tous les fichiers qui se situent dans un dossier diffusion sont accessibles en lecture à l’ensemble des utilisateurs authentifiés. Utilisez l’interface Mes Fichiers pour créer un dossier diffusion à la racine de votre bucket. Si tout a bien fonctionné, la commande Python ci-dessus devrait désormais afficher le chemin mon_nom_utilisateur_sspcloud/diffusion.
Plutôt que chaque membre du projet travaille avec ses propres fichiers sur son ordinateur, ce qui implique une synchronisation fréquente entre membres du groupe et limite la reproductibilité du fait des risques d’erreur, les fichiers sont mis sur un dépôt central, que chaque membre du groupe peut ensuite requêter.
Pour cela, il faut simplement s’accorder au sein du groupe pour utiliser le bucket d’un des membres du projet, et s’assurer que les autres membres du groupe peuvent accéder aux données, en les mettant dans le dossier diffusion du bucket choisi.
3.4.2 Récupération et stockage de données
Maintenant que nous savons où mettre nos données sur MinIO, regardons comment le faire en pratique depuis Python.
Cas d’un Dataframe
Reprenons un exemple issu du cours sur les API pour simuler une étape de récupération de données coûteuse en temps.
import requests
import pandas as pd
url_api = "https://koumoul.com/data-fair/api/v1/datasets/dpe-france/lines?format=json&q_mode=simple&qs=code_insee_commune_actualise%3A%2201450%22&size=100&select=%2A&sampling=neighbors"
response_json = requests.get(url_api).json()
df_dpe = pd.json_normalize(response_json["results"])
df_dpe.head(2)| classe_consommation_energie | tr001_modele_dpe_type_libelle | annee_construction | _geopoint | latitude | surface_thermique_lot | numero_dpe | _i | tr002_type_batiment_description | geo_adresse | ... | geo_score | classe_estimation_ges | nom_methode_dpe | tv016_departement_code | consommation_energie | date_etablissement_dpe | longitude | _score | _id | version_methode_dpe | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | E | Vente | 1 | 45.927488,5.230195 | 45.927488 | 106.87 | 1301V2000001S | 2 | Maison Individuelle | Rue du Chateau 01800 Villieu-Loyes-Mollon | ... | 0.58 | B | Méthode Facture | 01 | 286.0 | 2013-04-15 | 5.230195 | None | HJt4TdUa1W0wZiNoQkskk | NaN |
| 1 | G | Vente | 1960 | 45.931376,5.230461 | 45.931376 | 70.78 | 1301V1000010R | 9 | Maison Individuelle | 552 Rue Royale 01800 Villieu-Loyes-Mollon | ... | 0.34 | D | Méthode 3CL | 01 | 507.0 | 2013-04-22 | 5.230461 | None | UhMxzza1hsUo0syBh9DxH | 3CL-DPE, version 1.3 |
2 rows × 23 columns
Cette requête nous permet de récupérer un DataFrame Pandas, dont les deux premières lignes sont imprimées ci-dessus. Dans notre cas le processus est volontairement simpliste, mais on peut imaginer que de nombreuses étapes de requêtage / préparation de la données sont nécessaires pour aboutir à un dataframe exploitable dans la suite du projet, et que ce processus est coûteux en temps. On va donc stocker ces données “intermédiaires” sur MinIO afin de pouvoir exécuter la suite du projet sans devoir refaire tourner tout le code qui les a produites.
On peut utiliser les fonctions d’export de Pandas, qui permettent d’exporter dans différents formats de données. Vu qu’on est dans le cloud, une étape supplémentaire est nécessaire : on ouvre une connexion vers MinIO, puis on exporte notre dataframe.
MY_BUCKET = "mon_nom_utilisateur_sspcloud"
FILE_PATH_OUT_S3 = f"{MY_BUCKET}/diffusion/df_dpe.csv"
with fs.open(FILE_PATH_OUT_S3, 'w') as file_out:
df_dpe.to_csv(file_out)On peut vérifier que notre fichier a bien été uploadé via l’interface Mes Fichiers ou bien directement en Python en interrogeant le contenu du dossier diffusion de notre bucket :
fs.ls(f"{MY_BUCKET}/diffusion")On pourrait tout aussi simplement exporter notre dataset en Parquet, pour limiter l’espace de stockage et maximiser les performances à la lecture. Attention : vu que Parquet est un format compressé, il faut préciser qu’on écrit un fichier binaire : le mode d’ouverture du fichier passé à la fonction fs.open passe de w (write) à wb (write binary).
FILE_PATH_OUT_S3 = f"{MY_BUCKET}/diffusion/df_dpe.parquet"
with fs.open(FILE_PATH_OUT_S3, 'wb') as file_out:
df_dpe.to_parquet(file_out)Cas de fichiers
Dans la partie précédente, on était dans le cas “simple” d’un dataframe, ce qui nous permettait d’utiliser directement les fonctions d’export de Pandas. Maintenant, imaginons qu’on ait plusieurs fichiers d’entrée, pouvant chacun avoir des formats différents. Un cas typique de tels fichiers sont les fichiers ShapeFile, qui sont des fichiers de données géographiques, et se présentent sous forme d’une combinaison de fichiers (cf. chapitre sur GeoPandas). Commençons par récupérer un fichier .shp pour voir sa structure.
On récupère ci-dessous les contours du département de la Réunion produits par l’IGN, sous la forme d’une archive .7z qu’on va décompresser en local dans un dossier departements_fr.
import io
import os
import requests
import py7zr
# Import et décompression
contours_url = "https://data.geopf.fr/telechargement/download/ADMIN-EXPRESS-COG-CARTO/ADMIN-EXPRESS-COG-CARTO_3-2__SHP_RGR92UTM40S_REU_2023-05-03/ADMIN-EXPRESS-COG-CARTO_3-2__SHP_RGR92UTM40S_REU_2023-05-03.7z"
response = requests.get(contours_url, stream=True)
with py7zr.SevenZipFile(io.BytesIO(response.content), mode='r') as archive:
archive.extractall(path="departements_fr")
# Vérification du dossier (local, pas sur S3)
os.listdir("departements_fr")['ADMIN-EXPRESS-COG-CARTO_3-2__SHP_RGR92UTM40S_REU_2023-05-03']Vu qu’il s’agit cette fois de fichiers locaux et non d’un dataframe Pandas, on doit utiliser le package s3fs pour transférer les fichiers du filesystem local au filesystem distant (MinIO). Grâce à la commande put, on peut copier en une seule commande le dossier sur MinIO. Attention à bien spécifier le paramètre recursive=True, qui permet de copier à la fois un dossier et son contenu.
fs.put("departements_fr/", f"{MY_BUCKET}/diffusion/departements_fr/", recursive=True)Vérifions que le dossier a bien été copié :
fs.ls(f"{MY_BUCKET}/diffusion/departements_fr")Si le dossier, comme ici, contient des fichiers à plusieurs niveaux, il faudra parcourir la liste de manière récursive pour accéder aux fichiers. Si par exemple, on s’intéresse exclusivement aux découpages des communes, on pourra par exemple utiliser un glob:
fs.glob(f'{MY_BUCKET}/diffusion/departements_fr/**/COMMUNE.*')Si tout a bien fonctionné, la commande ci-dessus devrait renvoyer une liste contenant les chemins sur MinIO des différents fichiers (.shp, .shx, .prj, etc.) constitutifs du ShapeFile des communes de la Réunion.
3.4.3 Utilisation des données
En sens inverse, pour récupérer les fichiers depuis MinIO dans une session Python, les commandes sont symétriques.
Cas d’un dataframe
Attention à bien passer cette fois le paramètre r (read, pour lecture) et non plus w (write, pour écriture) à la fonction fs.open afin de ne pas écraser le fichier !
MY_BUCKET = "mon_nom_utilisateur_sspcloud"
FILE_PATH_S3 = f"{MY_BUCKET}/diffusion/df_dpe.csv"
# Import
with fs.open(FILE_PATH_S3, 'r') as file_in:
df_dpe = pd.read_csv(file_in)
# Vérification
df_dpe.head(2)De même, si le fichier est en Parquet (en n’oubliant pas de passer de r à rb pour tenir compte de la compression) :
MY_BUCKET = "mon_nom_utilisateur_sspcloud"
FILE_PATH_S3 = f"{MY_BUCKET}/diffusion/df_dpe.parquet"
# Import
with fs.open(FILE_PATH_S3, 'rb') as file_in:
df_dpe = pd.read_parquet(file_in)
# Vérification
df_dpe.head(2)Cas de fichiers
Dans le cas de fichiers, on va devoir dans un premier temps rapatrier les fichiers de MinIO vers la machine local (en l’occurence, le service ouvert sur le SSP Cloud).
# Récupération des fichiers depuis MinIO vers la machine locale
fs.get(f"{MY_BUCKET}/diffusion/departements_fr/", "departements_fr/", recursive=True)Puis on les importe classiquement depuis Python avec le package approprié. Dans le cas des ShapeFile, où les différents fichiers sont en fait des parties d’un seul et même fichier, une seule commande permet de les importer après les avoir rappatriés.
- 1
-
Comme notre dossier contient beaucoup de fichiers, on va devoir faire une recherche dedans via, à nouveau, un
glob - 2
-
Pathlibnous donnant une liste, on se restreint au premier écho et on importe avecGeoPandas
3.5 Pour aller plus loin
Informations additionnelles
Ce site a été construit automatiquement par le biais d’une action Github utilisant le logiciel de publication reproductible Quarto
L’environnement utilisé pour obtenir les résultats est reproductible par le biais d’uv. Le fichier pyproject.toml utilisé pour construire cet environnement est disponible sur le dépôt linogaliana/python-datascientist
pyproject.toml
[project]
name = "python-datascientist"
version = "0.1.0"
description = "Source code for Lino Galiana's Python for data science course"
readme = "README.md"
requires-python = ">=3.13,<3.14"
dependencies = [
"altair>=6.0.0",
"black==24.8.0",
"cartiflette",
"contextily==1.6.2",
"duckdb>=0.10.1",
"folium>=0.19.6",
"gdal!=3.11.1",
"geoplot==0.5.1",
"graphviz==0.20.3",
"great-tables>=0.12.0",
"gt-extras>=0.0.8",
"ipykernel>=6.29.5",
"jupyter>=1.1.1",
"jupyter-cache==1.0.0",
"kaleido==0.2.1",
"langchain-community>=0.3.27",
"loguru==0.7.3",
"markdown>=3.8",
"nbclient==0.10.0",
"nbformat==5.10.4",
"nltk>=3.9.1",
"pip>=25.1.1",
"plotly>=6.1.2",
"plotnine>=0.15",
"polars==1.8.2",
"pyarrow>=17.0.0",
"pynsee==0.1.8",
"python-dotenv==1.0.1",
"python-frontmatter>=1.1.0",
"pywaffle==1.1.1",
"requests>=2.32.3",
"scikit-image==0.24.0",
"scipy>=1.13.0",
"selenium<4.39.0",
"spacy>=3.8.4",
"webdriver-manager==4.0.2",
"wordcloud==1.9.3",
]
[tool.uv.sources]
cartiflette = { git = "https://github.com/inseefrlab/cartiflette" }
gdal = [
{ index = "gdal-wheels", marker = "sys_platform == 'linux'" },
{ index = "geospatial_wheels", marker = "sys_platform == 'win32'" },
]
[[tool.uv.index]]
name = "geospatial_wheels"
url = "https://nathanjmcdougall.github.io/geospatial-wheels-index/"
explicit = true
[[tool.uv.index]]
name = "gdal-wheels"
url = "https://gitlab.com/api/v4/projects/61637378/packages/pypi/simple"
explicit = true
[dependency-groups]
dev = [
"nb-clean>=4.0.1",
]
Pour utiliser exactement le même environnement (version de Python et packages), se reporter à la documentation d’uv.
| SHA | Date | Author | Description |
|---|---|---|---|
| 60518fdb | 2025-08-20 15:45:42 | lgaliana | clean process |
| 73043ee7 | 2025-08-20 14:50:30 | Lino Galiana | retire l’historique inutile des données velib (#638) |
| c3d51646 | 2025-08-12 17:28:51 | Lino Galiana | Ajoute un résumé au début de chaque chapitre (première partie) (#634) |
| 94648290 | 2025-07-22 18:57:48 | Lino Galiana | Fix boxes now that it is better supported by jupyter (#628) |
| 7611f138 | 2025-06-18 13:43:09 | Lino Galiana | Traduction du chapitre S3 (#616) |
| e4a1e0bd | 2025-06-17 16:57:25 | lgaliana | commence la traduction du chapitre S3 |
| 78e4efe5 | 2025-06-17 12:40:57 | lgaliana | change variable that make pipeline fail |
| 14790b01 | 2025-06-16 16:57:32 | lgaliana | fix error api komoul |
| 8f8e6563 | 2025-06-16 15:42:32 | lgaliana | Les chapitres dans le bon ordre, ça serait mieux… |
| d8bacc63 | 2025-06-16 17:34:16 | Lino Galiana | Ménage de printemps: retire les vieux chapitres inutiles (#615) |
Notes de bas de page
Pour en savoir plus sur les enjeux liés à un choix de format de données, voir @dondon2023quels.↩︎
Il est recommandé de régulièrement consulter la documentation officielle de
pyarrowconcernant la lecture et écriture de fichiers et celle relative aux manipulations de données.↩︎Ce comportement est souvent rendu possible via des systèmes de fichiers virtuels ou des wrappers compatibles avec
pandas,pyarrow,duckdb, etc.↩︎
Citation
@book{galiana2025,
author = {Galiana, Lino},
title = {Python pour la data science},
date = {2025},
url = {https://pythonds.linogaliana.fr/},
doi = {10.5281/zenodo.8229676},
langid = {fr}
}



3.3 Comment faire avec Python ?
3.3.1 Les librairies principales
L’interaction entre ce système distant de fichiers et une session locale de Python est possible grâce à des API. Les deux principales librairies sont les suivantes :
Les librairies
pyarrowetduckdbque nous avons déjà présentées permettent également de traiter des données stockées sur le cloud comme si elles étaient sur le serveur local. C’est extrêmement pratique et permet de fiabiliser la lecture ou l’écriture de fichiers dans une architecture cloud.Sur le SSP Cloud, les jetons d’accès au stockage S3 sont injectés automatiquement dans les services lors de leur création. Ils sont ensuite valides pour une durée de 7 jours. Si l’icône du service passe du vert au rouge, cela signifie que ces jetons sont périmés, il faut donc sauvegarder son code / ses données et reprendre depuis un nouveau service.