!pip install duckdbCe chapitre présente la première application d’une journée de cours que j’ai donné à l’Université Dauphine dans le cadre des PSL Data Week.
Dérouler les _slides_ associées ci-dessous ou [cliquer ici](https://linogaliana.github.io/dauphine-week-data/#/title-slide) pour les afficher en plein écran.
Pour lire les données de manière efficace, nous
proposons d’utiliser le package duckdb.
Pour l’installer, voici la commande :
1 Pourquoi utiliser les pipelines ?
1.1 Définitions préalables
Ce chapitre nous amènera à explorer plusieurs écosystèmes, pour lesquels on retrouve quelques buzz-words dont voici les définitions :
| Terme | Définition |
|---|---|
| DevOps | Mouvement en ingénierie informatique et une pratique technique visant à l’unification du développement logiciel (dev) et de l’administration des infrastructures informatiques (ops) |
| MLOps | Ensemble de pratiques qui vise à déployer et maintenir des modèles de machine learning en production de manière fiable et efficace |
Ce chapitre fera des références régulières au cours de 3e année de l’ENSAE “Mise en production de projets data science”.
1.2 Objectif
Les chapitres précédents ont permis de montrer des bouts de code épars pour entraîner des modèles ou faire du preprocessing. Cette démarche est intéressante pour tâtonner mais risque d’être coûteuse ultérieurement s’il est nécessaire d’ajouter une étape de preprocessing ou de changer d’algorithme.
Les pipelines sont pensés pour simplifier la mise en production ultérieure d’un modèle de machine learning. Ils sont au coeur de la démarche de MLOps qui est présentée dans le cours de 3e année de l’ENSAE de “Mise en production de projets data science”, qui vise à simplifier la mise en oeuvre opérationnelle de projets utilisant des techniques de machine learning.
import matplotlib.pyplot as plt
import seaborn as sns
import numpy as np
import pandas as pd1.3 Les pipelines Scikit
Heureusement, Scikit propose un excellent outil pour proposer un cadre
général pour créer une chaîne de production machine learning. Il
s’agit des
pipelines.
Ils présentent de nombreux intérêts, parmi lesquels :
- Ils sont très pratiques et lisibles. On rentre des données en entrée, on n’appelle qu’une seule fois les méthodes
fitetpredictce qui permet de s’assurer une gestion cohérente des transformations de variables, par exemple après l’appel d’unStandardScaler; - La modularité rend aisée la mise à jour d’un pipeline et renforce la capacité à le réutiliser ;
- Ils permettent de facilement chercher les hyperparamètres d’un modèle. Sans pipeline, écrire un code qui fait du tuning d’hyperparamètres peut être pénible. Avec les pipelines, c’est une ligne de code ;
- La sécurité d’être certain que les étapes de preprocessing sont bien appliquées aux jeux de données désirés avant l’estimation.
Un des intérêts des pipelines scikit est qu’ils fonctionnent aussi avec
des méthodes qui ne sont pas issues de scikit.
Il est possible d’introduire un modèle de réseau de neurone Keras dans
un pipeline scikit.
Pour introduire un modèle économétrique statsmodels
c’est un peu plus coûteux mais nous allons proposer des exemples
qui peuvent servir de modèle et qui montrent que c’est faisable
sans trop de difficulté.
3 Un premier pipeline : random forest sur des variables standardisées
Notre premier pipeline va nous permettre d’intégrer ensemble:
- Une étape de preprocessing avec la standardisation de variables
- Une étape d’estimation du prix en utilisant un modèle de random forest
Pour le moment, on va prendre comme acquis un certain nombre de variables explicatives (les features) et les hyperparamètres du modèle.
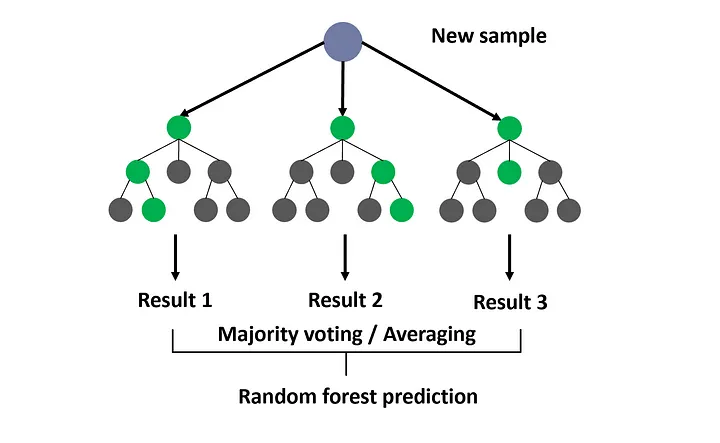
L’algorithme des random forest est une technique statistique basée sur les arbres de décision. Elle a été définie explicitement par l’un des pionniers du machine learning, Breiman (2001). Il s’agit d’une méthode ensembliste puisqu’elle consiste à utiliser plusieurs algorithmes (en l’occurrence des arbres de décision) pour obtenir une meilleure prédiction que ne le permettraient chaque modèle isolément.
Les random forest sont une méthode d’aggrégation1 d’arbres de décision. On calcule \(K\) arbres de décision et en tire, par une méthode d’agrégation, une règle de décision moyenne qu’on va appliquer pour tirer une prédiction de nos données.
L’un des intérêts des random forest est qu’il existe des méthodes pour déterminer l’importance relative de chaque variable dans la prédiction.
Nous allons ici partir d’un random forest avec des valeurs d’hyperparamètres données, à savoir la profondeur de l’arbre.
3.1 Définition des ensembles train et test
Nous allons donc nous restreindre à un sous-ensemble de colonnes dans un premier temps.
Nous allons également ne conserver que les transactions inférieures à 5 millions d’euros (on anticipe que celles ayant un montant supérieur sont des transactions exceptionnelles dont le mécanisme de fixation du prix diffère)
mutations2 = mutations.drop(
colonnes_surface.tolist() + ["Date mutation", "lprix"], # ajouter "confinement" si données 2020
axis = "columns"
).copy()
mutations2 = mutations2.loc[mutations2['Valeur fonciere'] < 5e6] #keep only values below 5 millions
mutations2.columns = mutations2.columns.str.replace(" ", "_")
mutations2 = mutations2.dropna(subset = ['dep','Code_type_local','month'])Notre pipeline va incorporer deux types de variables: les variables catégorielles et les variables numériques. Ces différents types vont bénéficier d’étapes de preprocessing différentes.
numeric_features = mutations2.columns[~mutations2.columns.isin(['dep','Code_type_local', 'month', 'Valeur_fonciere'])].tolist()
categorical_features = ['dep','Code_type_local','month']Au passage, nous avons abandonné la variable de code postal pour privilégier le département afin de réduire la dimension de notre jeu de données. Si on voulait vraiment avoir un bon modèle, il faudrait faire autrement car le code postal est probablement un très bon prédicteur du prix d’un bien, une fois que les caractéristiques du bien sont contrôlées.
Nous allons stratifier notre échantillonage de train/test par département afin de tenir compte, de manière minimale, de la géographie. Pour accélérer les calculs pour ce tutoriel, nous n’allons considérer que 30% des transactions observées sur chaque département.
Voici le code pour le faire:
mutations2 = mutations2.groupby('dep').sample(frac = 0.1, random_state = 123)Avec la fonction adéquate de Scikit, faire un découpage de mutations2
en train et test sets
en suivant les consignes suivantes:
- 20% des données dans l’échantillon de test ;
- L’échantillonnage est stratifié par départements ;
- Pour avoir des résultats reproductibles, choisir une racine égale à 123.
3.2 Définition du premier pipeline
Pour commencer, nous allons fixer la taille des arbres de décision avec
l’hyperparamètre max_depth = 2.
Notre pipeline va intégrer les étapes suivantes :
- Preprocessing :
- Les variables numériques vont être standardisées avec un
StandardScaler. Pour cela, nous allons utiliser la listenumeric_featuresdéfinie précédemment. - Les variables catégorielles vont être explosées avec un one hot encoding
(méthode
OneHotEncoderdescikit) Pour cela, nous allons utiliser la listecategorical_features
- Les variables numériques vont être standardisées avec un
- Random forest : nous allons appliquer l’estimateur ad hoc de
Scikit.
- Initialiser un random forest de profondeur 2. Fixer la racine à 123 pour avoir des résultats reproductibles.
- La première étape du pipeline (nommer cette couche preprocessor) consiste à appliquer les étapes de preprocessing adaptées à chaque type de variables:
- Pour les variables numériques, appliquer une étape d’imputation à la moyenne puis standardiser celles-ci
- Pour les variables catégorielles, appliquer un one hot encoding
- Appliquer comme couche de sortie le modèle défini plus tôt.
💡 Il est recommandé de s’aider de la documentation de Scikit. Si vous avez besoin d’un indice supplémentaire, consulter le pipeline présenté ci-dessous.
A l’issue de cet exercice, nous devrions obtenir le pipeline suivant.
Nous avons construit ce pipeline sous forme de couches successives. La couche
randomforest prendra automatiquement le résultat de la couche preprocessor
en input. La couche features permet d’introduire de manière relativement
simple (quand on a les bonnes méthodes) la complexité du preprocessing
sur données réelles dont les types divergent.
A cette étape, rien n’a encore été estimé. C’est très simple à mettre en oeuvre avec un pipeline.
- Estimer les paramètres du modèle sur le jeu d’entraînement
- Observer la manière dont les données d’entraînement sont transformées
par l’étape de preprocessing avec les méthodes adéquates sur 4 observations de
X_traintirées aléatoirement - Utiliser ce modèle pour prédire le prix sur l’échantillon de test. A partir de ces quelques prédictions, quel semble être le problème ?
- Observer la manière dont ce preprocessing peut s’appliquer sur deux exemples fictifs :
- Un appartement (
code_type_local = 2) dans le 75, vendu au mois de mai, unique lot de la vente avec 3 pièces, faisant 75m² ; - Une maison (
code_type_local = 1) dans le 06, vendue en décembre, dans une transaction avec 2 lots. La surface complète est de 180m² et le bien comporte 6 pièces.
- Un appartement (
- Déduire sur ces deux exemples le prix prédit par le modèle.
- Calculer et interpréter le RMSE sur l’échantillon de test. Ce modèle est-il satisfaisant ?
3.3 Variable importance
Les prédictions semblent avoir une assez faible variance, comme si des variables de seuils intervenaient. Nous allons donc devoir essayer de comprendre pourquoi.
La “variable importance” se réfère à la mesure de l’influence de chaque variable d’entrée sur la performance du modèle. L’impureté fait référence à l’incertitude ou à l’entropie présente dans un ensemble de données. Dans le contexte des random forest, cette mesure est souvent calculée en évaluant la réduction moyenne de l’impureté des nœuds de décision causée par une variable spécifique. Cette approche permet de quantifier l’importance des variables dans le processus de prise de décision du modèle, offrant ainsi des intuitions sur les caractéristiques les plus informatives pour la prédiction (plus de détails sur ce blog).
On ne va représenter, parmi notre ensemble important de colonnes, que celles qui ont une importance non nulle.
- Récupérer la feature importance directement depuis la couche adaptée de votre pipeline
- Utiliser le code suivant pour calculer l’intervalle de confiance de cette mesure d’importance:
std = np.std([tree.feature_importances_ for tree in pipe['randomforest'].estimators_], axis=0)- Représenter les variables d’importance non nulle. Qu’en concluez-vous ?
Le graphique d’importance des variables que vous devriez obtenir à l’issue de cet exercice est le suivant.
Les statistiques obtenues par le biais de cette variable importance sont un peu rudimentaires mais permettent déjà de comprendre le problème de notre modèle.
On voit donc que deux de nos variables déterminantes sont des effets fixes géographiques (qui servent à ajuster de la différence de prix entre Paris et les Hauts de Seine et le reste de la France), une autre variable est un effet fixe type de bien. Les deux variables qui pourraient introduire de la variabilité, à savoir la surface et, dans une moindre mesure, le nombre de lots, ont une importance moindre.
Idéalement, on utiliserait Yellowbrick pour représenter l’importance des variables
Mais en l’état actuel du pipeline on a beaucoup de variables dont le poids
est nul qui viennent polluer la visualisation. Vous pouvez
consulter la
documentation de Yellowbrick sur ce sujet
Les prédictions peuvent nous suggérer également qu’il y a un problème.
4 Restriction du champ du modèle
Mettre en oeuvre un bon modèle de prix au niveau France entière est complexe. Nous allons donc nous restreindre au champ suivant: les appartements dans Paris.
mutations_paris = mutations.drop(
colonnes_surface.tolist() + ["Date mutation", "lprix"], # ajouter "confinement" si données 2020
axis = "columns"
).copy()
mutations_paris = mutations_paris.loc[mutations_paris['Valeur fonciere'] < 5e6] #keep only values below 5 millions
mutations_paris.columns = mutations_paris.columns.str.replace(" ", "_")
mutations_paris = mutations_paris.dropna(subset = ['dep','Code_type_local','month'])
mutations_paris = mutations_paris.loc[mutations_paris['dep'] == "75"]
mutations_paris = mutations_paris.loc[mutations_paris['Code_type_local'] == 2].drop(['dep','Code_type_local'], axis = "columns")
mutations_paris.loc[mutations_paris['surface']>0]Reprendre les codes précédents et reconstruire notre pipeline sur la nouvelle base en mettant en oeuvre une méthode de boosting plutôt qu’une forêt aléatoire.
La correction de cet exercice est apparente pour simplifier les prochaines étapes mais essayez de faire celui-ci de vous-même.
A l’issue de cet exercice, vous devriez avoir des MDI proches de celles-ci :
from sklearn.ensemble import GradientBoostingRegressor
from sklearn.impute import SimpleImputer
from sklearn.preprocessing import StandardScaler, OneHotEncoder
from sklearn.pipeline import make_pipeline, Pipeline
from sklearn.compose import make_column_transformer
from sklearn.model_selection import train_test_split
mutations_paris = mutations.drop(
colonnes_surface.tolist() + ["Date mutation", "lprix"], # ajouter "confinement" si données 2020
axis = "columns"
).copy()
mutations_paris = mutations_paris.loc[mutations_paris['Valeur fonciere'] < 5e6] #keep only values below 5 millions
mutations_paris.columns = mutations_paris.columns.str.replace(" ", "_")
mutations_paris = mutations_paris.dropna(subset = ['dep','Code_type_local','month'])
mutations_paris = mutations_paris.loc[mutations_paris['dep'] == "75"]
mutations_paris = mutations_paris.loc[mutations_paris['Code_type_local'] == 2].drop(['dep','Code_type_local', 'Nombre_de_lots'], axis = "columns")
mutations_paris.loc[mutations_paris['surface']>0]
numeric_features = mutations_paris.columns[~mutations_paris.columns.isin(['month', 'Valeur_fonciere'])].tolist()
categorical_features = ['month']
reg = GradientBoostingRegressor(random_state=0)
numeric_pipeline = make_pipeline(
SimpleImputer(),
StandardScaler()
)
transformer = make_column_transformer(
(numeric_pipeline, numeric_features),
(OneHotEncoder(sparse_output = False, handle_unknown = "ignore"), categorical_features))
pipe = Pipeline(steps=[('preprocessor', transformer),
('boosting', reg)])
X_train, X_test, y_train, y_test = train_test_split(
mutations_paris.drop("Valeur_fonciere", axis = 1),
mutations_paris["Valeur_fonciere"],
test_size = 0.2, random_state = 123
)
pipe.fit(X_train, y_train)
pd.DataFrame(
pipe["boosting"].feature_importances_,
index = pipe[:-1].get_feature_names_out()
)5 Recherche des hyperparamètres optimaux avec une validation croisée
On détecte que le premier modèle n’est pas très bon et ne nous aidera pas vraiment à évaluer de manière fiable l’appartement de nos rêves.
On va essayer de voir si notre modèle ne serait pas meilleur avec des hyperparamètres plus adaptés. Après tout, nous avons choisi par défaut la profondeur de l’arbre mais c’était un choix au doigt mouillé.
❓️ Quels sont les hyperparamètres qu’on peut essayer d’optimiser ?
pipe['boosting'].get_params()Un détour par la documentation
nous aide à comprendre ceux sur lesquels on va jouer. Par exemple, il serait
absurde de jouer sur le paramètre random_state qui est la racine du générateur
pseudo-aléatoire.
X = pd.concat((X_train, X_test), axis=0)
Y = np.concatenate([y_train,y_test])Nous allons nous contenter de jouer sur les paramètres:
n_estimators: Le nombre d’arbres de décision que notre forêt contientmax_depth: La profondeur de chaque arbre
Il existe plusieurs manières de faire de la validation croisée. Nous allons ici
utiliser la grid search qui consiste à estimer et tester le modèle sur chaque
combinaison d’une grille de paramètres et sélectionner le couple de valeurs
des hyperparamètres amenant à la meilleure prédiction. Par défaut, scikit
effectue une 5-fold cross validation. Nous n’allons pas changer
ce comportement.
Comme expliqué précédemment, les paramètres s’appelent sous la forme
<step>__<parameter_name>
La validation croisée pouvant être très consommatrice de temps, nous
n’allons l’effectuer que sur un nombre réduit de valeurs de notre grille.
Il est possible de passer la liste des valeurs à passer au crible sous
forme de liste
(comme nous allons le proposer pour l’argument max_depth dans l’exercice ci-dessous) ou
sous forme d’array (comme nous allons le proposer pour l’argument n_estimators) ce qui est
souvent pratique pour générer un criblage d’un intervalle avec np.linspace.
Les estimations sont, par défaut, menées de manière séquentielle (l’une après
l’autre). Nous sommes cependant face à un problème
embarassingly parallel.
Pour gagner en performance, il est recommandé d’utiliser l’argument
n_jobs=-1.
import numpy as np
from sklearn.model_selection import GridSearchCV
import time
start_time = time.time()
# Parameters of pipelines can be set using ‘__’ separated parameter names:
param_grid = {
"boosting__n_estimators": np.linspace(5,25, 5).astype(int),
"boosting__max_depth": [2,4]
}
grid_search = GridSearchCV(pipe, param_grid=param_grid)
grid_search.fit(X_train, y_train)
end_time = time.time()
print(f"Elapsed time : {int(end_time - start_time)} seconds")grid_search.best_params_
grid_search.best_estimator_Toutes les performances sur les ensembles d’échantillons et de test sur la grille d’hyperparamètres sont disponibles dans l’attribut:
perf_random_forest = pd.DataFrame(grid_search.cv_results_)Regardons les résultats moyens pour chaque valeur des hyperparamètres:
fig, ax = plt.subplots(1)
g = sns.lineplot(data = perf_random_forest, ax = ax,
x = "param_boosting__n_estimators",
y = "mean_test_score",
hue = "param_boosting__max_depth")
g.set(xlabel='Number of estimators', ylabel='Mean score on test sample')
g
plt.legend(bbox_to_anchor=(1.05, 1), loc='upper left', borderaxespad=0,
title='Depth of trees')Globalement, à profondeur d’arbre donnée, le nombre d’arbres affecte la performance. Changer la profondeur de l’arbre améliore la performance de manière plus marquée.
Maintenant, il nous reste à re-entraîner le modèle avec ces nouveaux paramètres sur l’ensemble du jeu de train et l’évaluer sur l’ensemble du jeu de test :
pipe_optimal = grid_search.best_estimator_
pipe_optimal.fit(X_train, y_train)
compar = pd.DataFrame([y_test, pipe_optimal.predict(X_test)]).T
compar.columns = ['obs','pred']
compar['diff'] = compar.obs - compar.predOn obtient le RMSE suivant :
Et si on regarde la qualité en prédiction:
On obtient plus de variance dans la prédiction, c’est déjà un peu mieux. Cependant, cela reste décevant pour plusieurs raisons:
- nous n’avons pas fait d’étape de sélection de variable
- nous n’avons pas chercher à déterminer si la variable à prédire la plus pertinente était le prix ou une transformation de celle-ci (par exemple le prix au \(m^2\))
6 Prochaine étape
Nous avons un modèle certes perfectible mais fonctionnel. La question qui se pose maintenant c’est d’essayer d’en faire quelque chose au service des utilisateurs. Cela nous amène vers la question de la mise en production.
Ceci est l’objet du prochain chapitre. Il s’agira d’une version introductive des enjeux évoqués dans le cadre du cours de 3e année de mise en production de projets de data science.
Références
Informations additionnelles
Ce site a été construit automatiquement par le biais d’une action Github utilisant le logiciel de publication reproductible Quarto
L’environnement utilisé pour obtenir les résultats est reproductible par le biais d’uv. Le fichier pyproject.toml utilisé pour construire cet environnement est disponible sur le dépôt linogaliana/python-datascientist
pyproject.toml
[project]
name = "python-datascientist"
version = "0.1.0"
description = "Source code for Lino Galiana's Python for data science course"
readme = "README.md"
requires-python = ">=3.13,<3.14"
dependencies = [
"altair>=6.0.0",
"black==24.8.0",
"cartiflette",
"contextily==1.6.2",
"duckdb>=0.10.1",
"folium>=0.19.6",
"gdal!=3.11.1",
"geoplot==0.5.1",
"graphviz==0.20.3",
"great-tables>=0.12.0",
"gt-extras>=0.0.8",
"ipykernel>=6.29.5",
"jupyter>=1.1.1",
"jupyter-cache==1.0.0",
"kaleido==0.2.1",
"langchain-community>=0.3.27",
"loguru==0.7.3",
"markdown>=3.8",
"nbclient==0.10.0",
"nbformat==5.10.4",
"nltk>=3.9.1",
"pip>=25.1.1",
"plotly>=6.1.2",
"plotnine>=0.15",
"polars==1.8.2",
"pyarrow>=17.0.0",
"pynsee==0.1.8",
"python-dotenv==1.0.1",
"python-frontmatter>=1.1.0",
"pywaffle==1.1.1",
"requests>=2.32.3",
"scikit-image==0.24.0",
"scipy>=1.13.0",
"selenium<4.39.0",
"spacy>=3.8.4",
"webdriver-manager==4.0.2",
"wordcloud==1.9.3",
]
[tool.uv.sources]
cartiflette = { git = "https://github.com/inseefrlab/cartiflette" }
gdal = [
{ index = "gdal-wheels", marker = "sys_platform == 'linux'" },
{ index = "geospatial_wheels", marker = "sys_platform == 'win32'" },
]
[[tool.uv.index]]
name = "geospatial_wheels"
url = "https://nathanjmcdougall.github.io/geospatial-wheels-index/"
explicit = true
[[tool.uv.index]]
name = "gdal-wheels"
url = "https://gitlab.com/api/v4/projects/61637378/packages/pypi/simple"
explicit = true
[dependency-groups]
dev = [
"nb-clean>=4.0.1",
]
Pour utiliser exactement le même environnement (version de Python et packages), se reporter à la documentation d’uv.
| SHA | Date | Author | Description |
|---|---|---|---|
| fe573ec0 | 2025-12-23 12:54:11 | Lino Galiana | Un syllabus sous la forme d’un joli tableau (#667) |
| d56f6e9e | 2025-11-25 08:30:59 | lgaliana | Un petit coup de neuf sur les consignes et corrections pandas related |
| 3e04ed3c | 2025-11-24 20:08:05 | lgaliana | Avoid deprecated ravel method and fix notebooks launch url |
| 94648290 | 2025-07-22 18:57:48 | Lino Galiana | Fix boxes now that it is better supported by jupyter (#628) |
| 91431fa2 | 2025-06-09 17:08:00 | Lino Galiana | Improve homepage hero banner (#612) |
| 80fa8152 | 2025-01-31 20:10:50 | Lino Galiana | Mise en forme notebook colab (#593) |
| 5ff770b5 | 2024-12-04 10:07:34 | lgaliana | Partie ML plus esthétique |
| d2422572 | 2024-08-22 18:51:51 | Lino Galiana | At this point, notebooks should now all be functional ! (#547) |
| c641de05 | 2024-08-22 11:37:13 | Lino Galiana | A series of fix for notebooks that were bugging (#545) |
| 0908656f | 2024-08-20 16:30:39 | Lino Galiana | English sidebar (#542) |
| 8f0d2e16 | 2024-05-07 15:06:45 | lgaliana | Duplicate data from datagouv |
| c9f9f8a7 | 2024-04-24 15:09:35 | Lino Galiana | Dark mode and CSS improvements (#494) |
| 06d003a1 | 2024-04-23 10:09:22 | Lino Galiana | Continue la restructuration des sous-parties (#492) |
| 8c316d0a | 2024-04-05 19:00:59 | Lino Galiana | Fix cartiflette deprecated snippets (#487) |
| 005d89b8 | 2023-12-20 17:23:04 | Lino Galiana | Finalise l’affichage des statistiques Git (#478) |
| 3fba6124 | 2023-12-17 18:16:42 | Lino Galiana | Remove some badges from python (#476) |
| 4cd44f35 | 2023-12-11 17:37:50 | Antoine Palazzolo | Relecture NLP (#474) |
| 16842200 | 2023-12-02 12:06:40 | Antoine Palazzolo | Première partie de relecture de fin du cours (#467) |
| e4642eeb | 2023-11-27 17:02:05 | Lino Galiana | Deploy ML model as API (#460) |
| a06a2689 | 2023-11-23 18:23:28 | Antoine Palazzolo | 2ème relectures chapitres ML (#457) |
| 4960f2b7 | 2023-11-22 12:02:32 | Lino Galiana | Chapitre pipeline scikit sur DVF (#454) |
| 69cf52bd | 2023-11-21 16:12:37 | Antoine Palazzolo | [On-going] Suggestions chapitres modélisation (#452) |
| 889a71ba | 2023-11-10 11:40:51 | Antoine Palazzolo | Modification TP 3 (#443) |
| 652009df | 2023-10-09 13:56:34 | Lino Galiana | Finalise le cleaning (#430) |
| a7711832 | 2023-10-09 11:27:45 | Antoine Palazzolo | Relecture TD2 par Antoine (#418) |
| 154f09e4 | 2023-09-26 14:59:11 | Antoine Palazzolo | Des typos corrigées par Antoine (#411) |
| 3bdf3b06 | 2023-08-25 11:23:02 | Lino Galiana | Simplification de la structure 🤓 (#393) |
| 78ea2cbd | 2023-07-20 20:27:31 | Lino Galiana | Change titles levels (#381) |
| 29ff3f58 | 2023-07-07 14:17:53 | linogaliana | description everywhere |
| f21a24d3 | 2023-07-02 10:58:15 | Lino Galiana | Pipeline Quarto & Pages 🚀 (#365) |
| 38693f62 | 2023-04-19 17:22:36 | Lino Galiana | Rebuild visualisation part (#357) |
| 32486330 | 2023-02-18 13:11:52 | Lino Galiana | Shortcode rawhtml (#354) |
| f10815b5 | 2022-08-25 16:00:03 | Lino Galiana | Notebooks should now look more beautiful (#260) |
| 494a85ae | 2022-08-05 14:49:56 | Lino Galiana | Images featured ✨ (#252) |
| d201e3cd | 2022-08-03 15:50:34 | Lino Galiana | Pimp la homepage ✨ (#249) |
| 12965bac | 2022-05-25 15:53:27 | Lino Galiana | :launch: Bascule vers quarto (#226) |
| 9c71d6e7 | 2022-03-08 10:34:26 | Lino Galiana | Plus d’éléments sur S3 (#218) |
| ed2ddec8 | 2022-01-04 13:32:43 | Expressso | retire typo (#212) |
| 66e2837c | 2021-12-24 16:54:45 | Lino Galiana | Fix a few typos in the new pipeline tutorial (#208) |
| e94c1c5b | 2021-12-23 21:34:46 | Lino Galiana | Un tutoriel sur les pipelines :tada: (#203) |
Les références
Notes de bas de page
Les random forest sont l’une des principales méthodes ensemblistes. Outre cette approche, les plus connues sont le bagging (boostrap aggregating) et le boosting qui consistent à choisir la prédiction à privilégier selon des algorithmes de choix différens. Par exemple le bagging est une technique basée sur le vote majoritaire (Breiman 1996). Cette technique s’inspire du bootstrap qui, en économétrie, consiste à ré-estimer sur K sous-échantillons aléatoires des données un estimateur afin d’en tirer, par exemple, un intervalle de confiance empirique à 95%. Le principe du bagging est le même. On ré-estime K fois notre estimateur (par exemple un arbre de décision) et propose une règle d’agrégation pour en tirer une règle moyennisée et donc une prédiction. Le boosting fonctionne selon un principe différent, basé sur l’optimisation de combinaisons de classifieurs faibles.↩︎
Citation
@book{galiana2025,
author = {Galiana, Lino},
title = {Python pour la data science},
date = {2025},
url = {https://pythonds.linogaliana.fr/},
doi = {10.5281/zenodo.8229676},
langid = {fr}
}


2 Comment créer un pipeline
Un pipeline est un enchaînement d’opérations qu’on code en enchainant des pairs (clé, valeur) :
transformet éventuellement une transformation inverse).Au sein d’une étape de pipeline, les paramètres d’un estimateur sont accessibles avec la notation
<estimator>__<parameter>. Cela permet de fixer des valeurs pour les arguments des fonctionsscikitqui sont appelées au sein d’un pipeline. C’est cela qui rendra l’approche des pipelines particulièrement utile pour la grid search :Ces pipelines sont initialisés sans données, il s’agit d’une structure formelle que nous allons ensuite ajuster en entraînant des modèles.
2.1 Données utilisées
Nous allons utiliser les données de transactions immobilières DVF pour chercher la meilleure manière de prédire, sachant les caractéristiques d’un bien, son prix.
Ces données sont mises à disposition sur
data.gouv. Néanmoins, le format csv n’étant pas pratique pour importer des jeux de données volumineux, nous proposons de privilégier la versionParquetmise à disposition par Eric Mauvière surdata.gouv. L’approche la plus efficace pour lire ces données est d’utiliserDuckDBafin de lire le fichier, extraire les colonnes d’intérêt puis passer àPandas(pour en savoir plus sur l’intérêt deDuckDBpour lire des fichiers volumineux, vous pouvez consulter ce post de blog ou celui-ci écrit par Eric Mauvière).Même si, en soi, les gains de temps sont faibles car
DuckDBoptimise les requêtes HTTPS nécessaires à l’import des données, nous proposons de télécharger les données pour réduire les besoins de bande passante.En premier lieu, puisque cela va faciliter les requêtes SQL ultérieures, on crée une vue :
Les données prennent la forme suivante :
Les variables que nous allons conserver sont les suivantes, nous allons les reformater pour la suite de l’exercice.
Le fichier
Parquetmis à disposition surdata.gouvprésente une incohérence de mise en forme de certaines colonnes à cause des virgules qui empêchent le formattage sous forme de colonne numérique.Le code ci-dessus effectue la conversion adéquate au niveau de
Pandas.Introduire un effet confinement
Si vous travaillez avec les données de 2020, n’oubliez pas d’intégrer l’effet confinement dans vos modèles puisque cela a lourdement affecté les possibilités de transaction sur cette période, donc l’effet potentiel de certaines variables explicatives du prix.
Pour introduire cet effet, vous pouvez créer une variable indicatrice entre les dates en question:
Comme nous travaillons sur les données de 2022, nous pouvons nous passer de cette variable.
Les données DVF proposent une observation par transaction. Ces transactions peuvent concerner plusieurs lots. Par exemple, un appartement avec garage et cave comportera trois lots.
Pour simplifier, on va créer une variable de surface qui agrège les différentes informations de surface disponibles dans le jeu de données. Les agréger revient à supposer que le modèle de fixation des prix est le même entre chaque lot. C’est une hypothèse simplificatrice qu’une personne plus experte du marché immobilier, ou qu’une approche propre de sélection de variable pourrait amener à nier. En effet, les variables en question sont faiblement corrélées les unes entre elles, à quelques exceptions près (Figure 2.1):